Gruppenseite - KWS
Diese Seite enthält die gesammelten Materialien der Gruppe 6 - KWS für die Portfolioprüfung.
Teilnehmer-innen
- Wollowski, Nicola
- Schneider, Lena
- Kessel, Laurin
Diskrete Modellierung: SIR Modell mit Austauschprozess
Unser Ziel war es, die Ausbreitung von COVID-19 mittels des modifizierten SIR-Modells in einem Gebiet in Deutschland zu modellieren. Dazu wurde das Gebiet zunächst in einzelne Gitterpunkte diskretisiert. Mittels eines Austauschprozesses wurde innerhalb des Modells die Bewegung der Menschen implemetiert und mit einem Kompartimentmodell die Ansteckung simuliert. Ein Zeitschritt bestand demnach aus zwei Teilschritten:
1. Transportprozess: Bewegung der Menschen von einer Zelle in ihre Nachbarzellen 2. Epidemiologischer Prozess: Durchführung des SIR Modells innerhalb der Zelle
Das modifizierte SIR Modell
Im Fall, dass die gestorbenen Infizierten nicht in der Gruppe der Genesenen - R erfasst werden sollen, kann das SIR Modell in eine andere Form überführt werden. Die Differentialgleichungen sehen dabei wie folgt aus:
hier ist die Infektionsrate aus dem SI Modell, bezeichnet die konstate Sterberate mit der die Infizierten aus der Gesamtpopulation
ausscheiden, beschreibt die Wechselrate zwischen der Gruppe der 'Infected' zu der Gruppe der 'Recovered' , beschreibt die Population der Genesenen (ohne Tote) und einen konstanten Faktor, der den prozentualen Anteil der durch Tests erfassten Infizierten beschreibt.
Die diskretisierte Form der DGLs lautet wie folgt:
In unserem Programm wurden S,I,R und D genau so programmiert. Mit der Schrittweite [in Tagen] erhalten wir:
Succeptible function wert=S_funktion(j,i,t,r,w,d,S,HS,HI,HR,HD) wert = HS(j,i)-slowdown2(t-1)/((HS(j,i)+HI(j,i)+HR(j,i))*r)*HS(j,i)*HI(j,i); endfunction Infected function wert=I_funktion(j,i,t,r,w,d,S,HS,HI,HR,HD) wert = HI(j,i)+slowdown2(j,i)/((HS(j,i)+HI(j,i)+HR(j,i)))*HS(j,i)*HI(j,i)-w*HI(j,i); endfunction Recovered function wert=R_funktion(j,i,t,r,w,d,NM,HS,HI,HR,HD) wert = HR(j,i)+w*HI(j,i)-d*HI(j,i); endfunction Dead function wert=D_funktion(j,i,t,r,w,d,NM,HS,HI,HR,HD) wert = HD(j,i)+d*HI(j,i); endfunction
HX sind hierbei Hilfsmatritzen für S,I,R und D, mit denen die Berechnung des Kompartimentmodells durchgeführt wird. In jeder Zelle kann die Kapazität beschrieben werden als . steht hier für die Infektionsrate abhängig von t, wobei t=0 dem 24. Februar 2020 entspricht.
Infektionsrate

Die Infektionsrate berechnet sich aus der Infiziertenzahl wie folgt:
wurde über eine polynomialen Regression 4. Grades aus den täglich veröffentlichten Daten des Robert Koch Instituts ermittelt (also: [Tag]).
#--Aktuelle RKI Daten auslesen--#
A=coronaData_aktuell();
inf_falleWHO=A(1,:);
inf_falleWHOrecovered=A(3,:);
inf_falleWHOtoten=A(2,:);
inf_falleWHOaktuell=inf_falleWHO-inf_falleWHOrecovered;
n=length(inf_falleWHO);
timesWHO=[0:1:n-1];
#--Infektionsrate berechnen aus RKI-Daten--#
ccc(1)=0.5;
T=1
for i=T+1:n
ccc(i-T)=(inf_falleWHO(i)-inf_falleWHO(i-T))/(inf_falleWHO(i-T) );
endfor
#--Polynomiale Regression vierten Grades--#
p1 = polyfit (1:(n-1), ccc, 4)
p1y = polyval (p1, 1:(n-1))
a = p1(1)
b = p1(2)
c = p1(3)
d = p1(4)
e = p1(5)
#f = p1(6)
##--Graphische Ausgabe--##
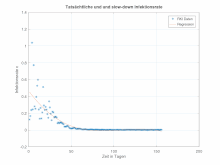
plot (1:(n-1),ccc,'*', 1:(n-1), p1y)
grid on
title ('Tatsächtliche und und slow-down Infektionsrate')
legend ('RKI Daten', 'Regression' )
xlabel('Zeit in Tagen')
ylabel('Infektionsrate c')
hold off
Da die Infektionsrate unter Anderem durch die Regression unter Null fällt, erhielten wir bei der Anwendung auf das SIR-Modell eine ungenaue Übereinstimmung mit den RKI-Daten. Daher haben wir die Infektionsrate per Hand angepasst.
Bevölkerungsdichte und -verteilung
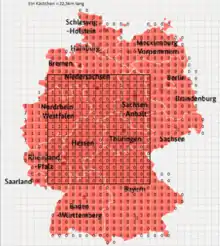
Die Bevölkerungsdichte aus Deutschland wurde für das von uns betrachtete Gebiet ermittelt. Dazu wurden zunächst die Daten der Bevölkerungsdichte (in 1000 Menschen pro qkm) der einzelnen Bundesländer von der Seite statista.de zusammengetragen. Außerdem wurden jeweils die Bevölkerungsdichten aus den Landeshauptstädten aus Wikipedia gesammelt. Diese Daten wurden anschließend mit Excel in einer Karte festgehalten, die Deutschland darstellt (1 Kästchen entspricht qkm). Das Rechteck mit dem schwarz markierten Rand, entspricht dem in dieser Arbeit simulierten Gebiet. Die ganzzahligen Werte in dem Bild sind darstellungsbedingt. Die tatsächlich erhaltenen Werte, sind in folgender Matrix dargestellt:
Zweidimensionale Bevölkerungsdichtematrix in 1000 pro qkm
Bd = [0.167 0.167 0.167 0.167 0.167 0.167 0.167 0.167 0.167 0.167 0.108 0.108 0.108 0.108 0.085;
0.167 0.167 0.167 0.167 0.167 0.167 2.63 2.63 0.167 0.167 0.167 0.108 0.108 0.108 0.108;
0.526 0.526 0.167 0.526 0.526 0.167 2.63 2.63 0.167 0.167 0.167 0.108 0.108 0.108 0.085;
0.526 0.526 0.526 0.526 0.526 0.526 0.167 0.167 0.167 0.167 0.108 0.108 0.108 0.108 0.108;
0.526 0.526 0.526 0.526 0.526 0.526 0.167 0.167 0.167 0.167 0.108 0.108 0.108 0.108 0.108;
0.526 0.526 0.526 0.526 0.526 0.526 0.167 0.167 0.167 0.132 0.132 0.108 0.108 0.108 0.108;
0.526 0.526 0.526 0.526 0.297 0.297 0.297 0.167 0.132 0.132 0.132 0.132 0.108 0.108 0.221;
0.526 0.526 0.526 0.526 0.297 0.297 0.297 0.297 0.132 0.132 0.793 0.793 0.108 0.108 0.221;
0.526 0.526 0.526 0.297 0.297 0.297 0.297 0.297 0.132 0.132 0.793 0.793 0.132 0.132 0.132;
0.206 0.206 0.297 0.297 0.297 0.297 0.297 0.297 0.132 0.132 0.132 0.132 0.132 0.132 0.132;
0.206 0.206 0.297 0.297 0.297 0.297 0.297 0.297 0.132 0.132 0.132 0.132 0.132 0.132 0.221;
0.206 0.206 3.074 0.297 0.297 0.297 0.297 0.185 0.185 0.185 0.185 0.185 0.185 0.185 0.185;
0.206 0.206 3.074 0.297 0.297 0.185 0.185 0.185 0.185 0.185 0.185 0.185 0.185 0.185 0.185;
0.206 0.206 2.236 0.297 0.297 0.185 0.185 0.185 0.185 0.185 0.185 0.185 0.185 0.185 0.185;
0.206 0.206 0.206 0.297 0.297 0.31 0.31 0.31 0.185 0.185 0.185 0.185 0.185 0.185 0.185;
0.206 0.206 0.206 0.31 0.31 0.31 0.31 0.31 0.185 0.185 0.185 0.185 0.185 0.185 0.185]
Die Bevölkerungsverteilung wurde aus der Bevölkerungsdichte abgeleitet, indem diese mit der zugehörigen Fläche multipliziert wurde.
Transportprozess
Der Transportprozess beruht auf einem Austausch von Menschen innerhalb der Moore-Nachbarschaft einer jeden Zelle. Wir gehen davon aus, dass der Anteil in jedem Zeitschritt in jeder Zelle gleichmäßig auf seine Nachbarn in der Moore-Nachbarschaft verteilt wird. Das betrachtete Gebiet erachten wir als abgeschlossen. Daher müssen wir berücksichtigen, dass Eckzellen lediglich 3 nächste Nachbarn haben, Kantenzellen 5 nächste Nachbarn und Zellen im inneren des Systems 8 nächste Nachbarn haben.
Um in der späteren Durchführung die Anzahl der nächsten Nachbarn einer jeder Zelle abrufen zu können, muss eine Nächste-Nachbar-Matrix erstellt werden, die dieselbe Dimension hat wie das Gebiet. Beispielhaft wird die Nächste-Nachbar-Matrix für und dargestellt:
Die Matrix mit den Einträgen steht hier stellvertretend für , , und . Der Verteilungsprozess einer Zelle auf seine umliegenden Nachbarzellen mit und lässt sich mathematisch wie folgt beschreiben:
Der Anteil bleibt in der Zelle zurück. Es gilt:
Dieser Transportprozess wird im Skript in folgenden Funktionen festgehalten:
Verteilung in die Nachbarzellen function wert=wandernder_anteil(l,k,j,i,q,t,NN,H,SIR) wert=H(l,k)+q/NN(j,i)*SIR(j,i,t-1); endfunction Verbleibender Anteil function wert=verbleibender_anteil(j,i,q,t,H,SIR) wert=H(j,i)+(1-q)*SIR(j,i,t-1); endfunction
Als Matrix kann man den Transport von einer Zelle (i,j) in seine 8nN auch wie folgt beschreiben:
Durchführung
Parameter SIR Modell
w = 1/14; #Wechselrate zu den Genesenen d = 0.003; #Todesrate r = 0.5; #Anteil der erfassten Infizierten T = 100; #Anzahl der Tage dt = [1:T]; #Zeitschritte
Parameter der räumlichen Ausbreitung
xend = 14; %in 22,5km #Seitenlänge x-Richtung in 22,5km
yend = 15; %in 22,5km #Seitenlänge y-Richtung in 22,5km
X = 40 ; #Anzahl der Gitterknoten in x-Richtung
hx = xend/(X-1); #Abstand zwischen zwei benachbarten Gitterknoten in x-Richtung
hy = hx; #Abstand zwischen zwei benachbarten Gitterknoten in y-Richtung
Y = floor((yend/hy))+1; #Anzahl der Gitterknoten in y-Richtung
h = hx;
[x,y] = meshgrid(0:hx:xend,0:hy:yend);
q = 0.5 ; #Anteil der pro Zeitschritt von einer Zelle in die nN wandert
Speichermatrizen und Hilfsmatrizen
#SIRD Matrizen erstellen S = zeros; #Speichermatrix Susceptible I = zeros(Y,X,T); #Speichermatrix Infected R = zeros(Y,X,T); #Speichermatrix Recovered D = zeros(Y,X,T); #Speichermatrix Dead #Hilfsmatrizen zum Zwischenspeichern HS = zeros(Y,X); #Hilfsmatrix Susceptible HI = zeros(Y,X); #Hilfsmatrix Infected HR = zeros(Y,X); #Hilfsmatrix Recovered HD = zeros(Y,X); #Hilfsmatrix Dead
Nächste-Nachbar-Matrix
NN = ones(Y,X)*8; #setzt erstmal jeden Eintrag gleich 8 #Kästchen in der Mitte haben 8nN #Randkästchen haben 5 nN for i=1:X NN(1,i)=5; NN(Y,i)=5; endfor for j=1:Y NN(j,1)=5; NN(j,X)=5; endfor #Eckkästchen haben 3 nN NN(1,1)=3; NN(1,X)=3; NN(Y,1)=3; NN(Y,X)=3;
Startkonfiguration erstellen
Die Susceptible-Verteilung entspricht im Zeitschritt der erstellten Bevölkerungsverteilung.
for i=1:X;
for j=1:Y;
S(j,i,1) = Bd(1+floor(j/X*yend),1+floor(i/Y*xend))*22.5^2*hx^2;
#Oder homogene Bevölkerungsverteilung
S(j,i,1) = b_mittel*hx^2;
endfor;
endfor;
S(:,:,1) = flipud(S(:,:,1));
Erste Infizierte
Wir betrachten die COVID-19-Ausbreitung ausgehend von 2 infizierten Personen, die am Frankfurter Flughafen das Gebiet betreten:
I(7,7,1) = 2/1000; #2 Infizierte Frankfurt Flughafen S(7,7,1) = S(7,7,1)-I(7,7,1);
Transport- & Reaktions-Schleife
#Alle Zeitschritte werden durchlaufen
for t=2:T
#ERSTENS: Menschenbewegung
t
#Pro Zeitschritt werden alle Felder durchlaufen
for i=1:X
for j=1:Y
#Pro Feld geht der Anteil q in die nN
for k=(i-1):(i+1)
for l=(j-1):(j+1)
#Liegt k oder l ausserhalb des Definitionsbereichs/ Schachbretts, soll nichts getan werden
if (((k==0 || k==X+1) || l==0) || l==Y+1)
a=0; #do nothing
#Der Anteil (1-q) bleibt in der Zelle selbst
elseif (i==k && j==l)
HS(l,k)=verbleibender_anteil(l,k,q,t,HS,S);
HI(l,k)=verbleibender_anteil(l,k,q,t,HI,I);
HR(l,k)=verbleibender_anteil(l,k,q,t,HR,R);
HD(l,k)=verbleibender_anteil(l,k,q,t,HD,D);
#Auf alle anderen nN wird der Anteil q aufgeteilt
elseif
HS(l,k)=wandernder_anteil(l,k,j,i,q,t,NN,HS,S);
HI(l,k)=wandernder_anteil(l,k,j,i,q,t,NN,HI,I);
HR(l,k)=wandernder_anteil(l,k,j,i,q,t,NN,HR,R);
HD(l,k)=wandernder_anteil(l,k,j,i,q,t,NN,HD,D);
endif
endfor
endfor
endfor
endfor
#ZWEITENS: SIR-Modell
for i=1:X
for j=1:Y
S(j,i,t) = S_funktion(j,i,t,r,w,d,S,HS,HI,HR,HD);
I(j,i,t) = I_funktion(j,i,t,r,w,d,S,HS,HI,HR,HD);
R(j,i,t) = R_funktion(j,i,t,r,w,d,NM,HS,HI,HR,HD);
D(j,i,t) = D_funktion(j,i,t,r,w,d,NM,HS,HI,HR,HD);
endfor
endfor
#Hilfsmatrix gleich Null setzen
HS = zeros(Y,X);
HI = zeros(Y,X);
HR = zeros(Y,X);
HD = zeros(Y,X);
endfor
Grafische Ausgabe und Abspeichern
for i=1:T/4
t=4*i
figure(t)
surfc(x',y',R(:,:,t), "EdgeColor", "none")
colormap ("jet")
colorbar
caxis ([0 max(max(max(R(:,:,:))))])
axis([1 xend 1 yend -0.1 max(max(max(R(:,:,:))))])
view(0,90)
xlabel("x")
ylabel("y")
test=["Fig_", num2str(t),".jpg"]
saveas(t, test)
endfor
Ergebnisse
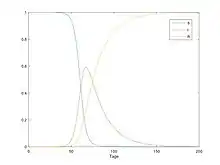
S, I, R und D, sowie die kumulierten Infizierten wurden für 100 Tage geplottet:
Succeptible:Infected:
Recovered:Dead: Infected kumuliert:
A1 Einfaches Kontaktmodell
Das Einfache Kontaktmodell dient dazu, den Einfluss der zufälligen Bewegung bzw. der Durchmischung der Bevölkerung auf die Verbreitung des Virus zu modellieren. Je nach Infektionsradius und Bewegungsgeschwindigkeit verlangsamt bzw. beschleunigt sich die Ausbreitung.
Durchführung
Startparameter festlegen
r = 1; #Infektionsradius v = 0.5; #Geschwindigkeit N = 100; #Anzahl Menschen T = 20; #Anzahl an Zeitschritten a = 15; #Größe des Quadrats P = zeros(3,N,T) #Positionsmatrix mit Zeitschritten; Drei Dimensionen × Zeit: x-Koordinate, y-Koordinate & Infiziert ja oder nein
Zufällige Startverteilung der Menschen festlegen
for i=1:N P(1,i,1) = a*rand(1); #Zufällige x-Koordinate im Intervall [0;a] P(2,i,1) = a*rand(1); #Zufällige y-Koordinate im Intervall [0;a] endfor
Erste infizierte Person generieren
patient_zero = randi(N) #Zufällige Person N würfeln P(3,patient_zero,1) = 1 #Person N als infiziert markieren
Plotten der Startverteilung
figure (1) hold on; plot(P(1,patient_zero,1),P(2,patient_zero,1), 'sr') ; plot(P(1,:,1),P(2,:,1), '*b') ; axis([-5 a+5 -5 a+5]);
Ausführen des Kontaktmodells
# Alle Zeitschritte durchlaufen
for t=2:T
#Neue Positionen zuteilen mittels Polarkoordinaten
for i=1:N
phi = rand(1)*2*pi; # zufälligen Winkel erzeugen, in dessen Richtung die Person läuft
vx = cos(phi); # Winkel in x-Richtung übersetzen
vy = sin(phi); # Winkel in y-Richtung übersetzen
P(1,i,t) = P(1,i,t-1)+vx*v; # neue x-Position zuweisen
P(2,i,t) = P(2,i,t-1)+vy*v; # neue y-Position zuweisen
endfor
#überprüfe ob neue Ansteckungen vorliegen
for i=1:N
if P(3,i,t-1)==1;
P(3,i,t) = 1; #Wenn Person im vorherigen Zeitpunkt infiziert, dann ist sie auch jetzt infiziert
#überprüfe nun, ob sich Person j im Ansteckungsradius von Person i befand
for j=1:N
xdistance = P(1,i,t-1)-P(1,j,t-1); #Distanz in x-Richtung
ydistance = P(2,i,t-1)-P(2,j,t-1); #Distanz in y-Richtung
distance = sqrt(xdistance^2+ydistance^2); #genormte Distanz mit Pythagoras
#Wenn Distanz kleiner gleich Ansteckungsradius, dann ist neue Person infiziert
if distance<=r
P(3,j,t)=1;
endif
endfor
endif
endfor
endfor
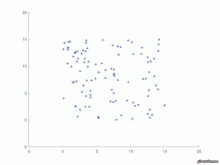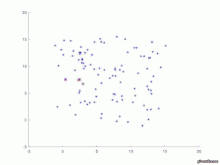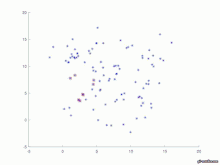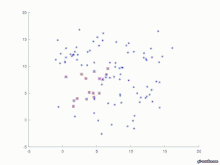
Graphische Ausgabe
for t=1:T figure (t+1) hold on; plot(P(1,:,t),P(2,:,t), '*b') ; for i=1:N if P(3,i,t)==1 plot(P(1,i,t),P(2,i,t), 'sr') ; endif endfor axis([-5 a+5 -5 a+5]); #test=["Fig_", num2str(t),".jpg"] #saveas(t, test) endfor
A2 Fundamentallösungen der Diffusionsgleichungen
Stationäre, homogene Diffusionsgleichung
für den 1-dimensionalen Fall
in
Spezialfall: c konstant --> Laplace - Gleichung:
bzw.
Fundamentallösung:
= Volumen der Einheitskugel in , = Vektornorm in
Für n=3 ist .
Durchführung
Gebiet erstellen
x = linspace(-5,5,201); #x-Koordinaten y = linspace(-5,5,201); #y-Koordinaten [X,Y] = meshgrid(x,y); #Koordinatenmatrix
Fundamentallösung implementieren
#für n=2 gilt: function wert=u(x,y) #Singularität rausschneiden if x==0 && y==0 wert = 1; else wert = -1/(2*pi)*log(sqrt(x.^2+y.^2)); endif endfunction
Graphische Ausgabe
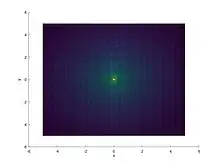
figure(1)
surface(X,Y,u(X,Y))
xlabel('x')
ylabel('y')
test=["Fig_", num2str(1),".jpg"]
saveas(1, test)
Instationäre, homogene Diffusionsgleichung
bzw.
, für c konstant
Fundamentallösung
= Quadrat der Euklidischen Norm von
Durchführung
Gebiet erstellen
x = linspace(-5,5,201); #x-Koordinaten y = linspace(-5,5,201); #y-Koordinaten [X,Y] = meshgrid(x,y); #Koordinatenmatrix
Parameter festlegen
a=0.08; #Diffusionskoeffizient T=20; #Zeitschritte
Fundamentallösung implementieren
for t=1:T u=@(x,y,t) 1/(4*pi*a*t)*exp(-(x.^2+y.^2)/(4*a*t));
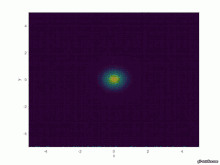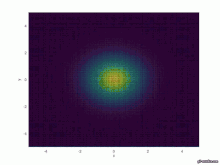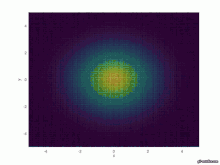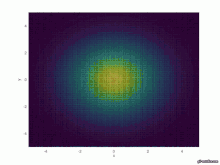
Graphische Ausgabe
figure(t)
surface(X,Y,u(X,Y,t))
axis([-5 5 -5 5 0 1])
xlabel('x')
ylabel('y')
test=["Fig_LI_", num2str(t),".jpg"]
saveas(t, test)
endfor
Stationäre, Inhomogene Diffusionsgleichung = Poissongleichung
Fundamentallösung:
, vergleiche | Fundamentallösung der homogenen, stationären DGL für n=2 mit verschobenem Argument
Durchführung
Gebiet erstellen
x = linspace(0,10,101); #x-Koordinaten y = linspace(0,10,101); #y-Koordinaten [X,Y] = meshgrid(x,y); #Koordinatenmatrix
Quellfunktion definieren
Um den Kreis mit dem Mittelpunkt (4,4) und dem Radius 0,5 soll die Quellfunktion den Wert 1 annehmen, ansonsten 0.
function wert=Quellfunktion(x,y) if sqrt((x.-4).^2+(y.-4).^2)<=0.5 wert=1; else wert=0; endif endfunction
Quellfunktion implementieren
for i=1:(length(X)) for j=1:(length(Y)) f(j,i)=1*Quellfunktion(X(j,i),Y(j,i)); endfor endfor
Fundamentallösung implementieren
for i=1:length(x) for j=1:length(y) #x-& y-star als Integrationsparameter bestimmen xstar = X(j,i)*ones(length(y), length(x)); ystar = Y(j,i)*ones(length(y), length(x)); phi = -1/(2*pi)*log(sqrt((xstar.-X).^2+(ystar.-Y).^2)); phi(j,i) = 0; #Singularitäten rausschneiden Int(j,i)=trapz(y,trapz(x,phi.*f,2)); endfor endfor
Graphische Ausgabe
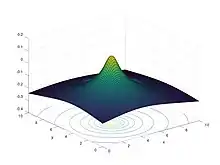
figure(1)
surfc(X,Y,Int)
xlabel('x')
ylabel('y')
test=["Fig_", num2str(1),".jpg"]
saveas(1, test)
Instationäre, homogene DGL mit Anfangswertproblem
Die Diffusiongleichung wird ergänzt durch eine Anfangsbedingung, die im Anfangszeitpunkt die Dichte der Infizierten beschreibt:
Fundamentallösung: , vergleiche Fundamentallösung der homogenen instationären Diffusionsgleichung mit verschobenem Argument
Durchführung
Gebiet-erstellen
x = linspace(0,10,101); y = linspace(0,10,101); [X,Y] = meshgrid(x,y);
a = 0.08; #Diffusionskoeffizient T = 10; #Zeitschritte
Anfangskonzentration
for i=1:(length(X)) for j=1:(length(Y)) u_null(j,i)=1*Quellfunktion(X(j,i),Y(j,i)); endfor endfor
Fundamentallösung implementieren
for k=1:T
t=k*0.5
for i=1:length(x)
for j=1:length(y)
xstar=X(j,i)*ones(length(y), length(x));
ystar=Y(j,i)*ones(length(y), length(x));
psi = 1/(4*pi*a*t)*exp(-sqrt((xstar-X).^2+(ystar-Y).^2)/(4*a*t));
Int(j,i)=trapz(y,trapz(x,psi.*u_null,2));
endfor
endfor
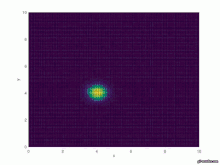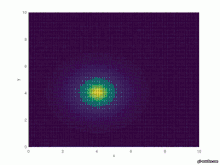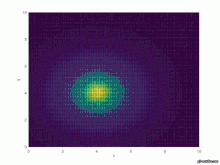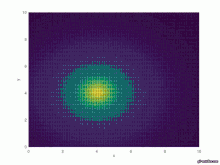
Graphische Ausgabe
figure(k)
surface(X,Y,Int)
#axis([0 10 0 10 0 3])
xlabel('x')
ylabel('y')
test=["Fig_", num2str(k),".jpg"]
saveas(k, test)
endfor
A3 Modifiziertes SIR Modell
Die inhomogene Diffusionsgleichung beschreibt zwei Prozesse, die miteinander gekoppelt sind:
1. der Reaktionsterm der rechten Seite beschreibt, wie der Stoff bzw. die Infizierten entstehen
2. die Diffusion beschreibt, wie sich der vorhandene Stoff bzw. die Infizierten im System ausbreiten.
In den folgenden Gleichungen entspricht der Reaktionsterm der rechten Seite der gewöhnlichen Differentialgleichung von .
Theoretischer Hintergrund
Exponentielles Wachstumsmodell
Ist die Bevölkerungsanzahl und damit die Kapazitätsgrenze unbeschränkt, findet ein exponentielles Wachstum der Infizierten statt.
= kummulative Anzahl der Infizierten zur Zeit , feste Ortskoordinate ,
= Anfangzustand der Infiziertenpopulation
Die Anzahl der Infizierten in neuem Zeitpunkt berechnet sich als:
= Infektionsrate, bezogen auf eine Zeiteinheit (Tag, Monat, Jahr)
Exponentielles Wachstumsmodell:
Lösung des exponentiellen Wachstumsmodells:
Logistisches Wachstumsmodell
Die Anzahl der Infizierten ist im logistischen Wachstumsmodell beschränkt, da sie nicht die Gesamtpopulation übersteigen kann. Deswegen verlangsamt sich das Populationswachstum, je weniger freie Kapazität (=Susceptibles) sich im System befindet.
Logistisches Wachstumsmodell:
Lösung des Modells:
Modifiziertes Kompartimentmodell
Das Kompartimentmodell teilt die Gesamtpopulation in mehrere Gruppen: S (Infektionsanfällige), I (Infizierte), R (Genesene) und D (Tote)
= Infektionsrate aus dem SI Modell,
= konstante Wechselrate, mit der die Infizierten in den Status der Genesenen oder Toten wechseln
= konstante Sterberate, mit der die Infizierten aus der Gesamtpopulation ausscheiden,
= konstanter Faktor, der den Prozentualanteil der durch Tests erfassten Infizierten beschreibt
Infektionsrate
Vergleiche die [| Infektionsrate der diskreten Modellierung des SIR-Modells].
Durchführung
Parameter des SIR Modells
NM = 2/3*83019.213 #Kapazitätsgrenze, 2/3 der deutschen Bevölkerung w = 1/14; #Wechselrate zu den Genesenen d = 0.003; #Todesrate r = 0.1; #Anteil der erfassten Infizierten (mittlerweile werden viel mehr erfasst r=r(t)) T = 156; #Anzahl der Tage (Länge von CoronaData Matrix) dt = [1:T]; delta_t = 0.01; #Schrittweite Tage
Anfangswerte festlegen
Ineu(1) = 16/1000; #Infizierte am Anfang Sneu(1) = NM-Ineu(1); Rneu(1) = 0; Tneu(1) = 0;
Ausführung des SIR-Modells
for t=1: floor (T/delta_t) #Modifiziertes SIR Modell Sneu(t+1) = Sneu(t)-delta_t*slowdown2(t*delta_t)/(NM*r)*Sneu(t)*Ineu(t); Ineu(t+1) = Ineu(t)+delta_t*slowdown2(t*delta_t)/(NM)*Sneu(t)*Ineu(t)-delta_t*w*Ineu(t); Rneu(t+1) = Rneu(t)+delta_t*w*Ineu(t)-delta_t*d*Ineu(t); Tneu(t+1) = Tneu(t)+delta_t*d*Ineu(t); endfor
Graphische Ausgabe
t=(1: floor (T/delta_t)+1)*delta_t;
figure(2)
plot( t, Ineu, t, Rneu, t, Tneu);
#plot( dt, Sneu, dt, Ineu, dt, Rneu, dt, Tneu);
#axis([0 T 0 NM])
xlabel('Tage')
hold on
#Vergleich mit RKI Daten
A=coronaData_aktuell();
inf_falleWHO=A(1,:);
inf_falleWHOrecovered=A(3,:);
inf_falleWHOtoten=A(2,:);
inf_falleWHOaktuell=inf_falleWHO-inf_falleWHOrecovered;
n=length(inf_falleWHO);
timesWHO=[0:1:n-1];
plot( timesWHO, inf_falleWHOaktuell/1000, 's', timesWHO,inf_falleWHOrecovered/1000, '*', timesWHO, inf_falleWHOtoten/1000, 'o');
legend( 'Ineu', 'Rneu', 'Tneu', 'Infizierte aktuell WHO', 'Erholt WHO', 'Tote WHO')
A4 FDM: Dirichlet und Neuman RWP
Theoretischer Hintergrund
In der Finiten Differenzen-Methode werden die Ableitungen der gesuchten Funktion durch Differenzenquotienten approximiert.
Taylorpolynom mit Lagrange'schem Restglied
Differenzenquotienten
- erster (vorwärts-) Differenzenquotient:
- erster (rückwärts-) Differenzenquotient:
- erster zentraler Differenzenquotient:
- Zweiter Differenzenquotient:
Randwertproblem
Das Randwertproblem beschreibt den Diffusionsprozess auf einem beschränkten Gebiet . Die Randwertbedingungen (z.B. Dirichlet oder Neumann) definieren dabei das Verhalten der unbekannten Funktion am Rand des Gebiets . Hier wird die stationäre, inhomogene Poissongleichung betrachtet:
Dirichlet-Randbedingungen
Wird die gesuchte Funktion am Rand durch eine gegebene Funktion definiert, erfüllt die Dirichlet-Randbedingung:
Poisson-Gleichung mit Dirichlet-Randwertproblem
Sei u die gesuchte Funktion definiert auf einem Rechteck D: . Wir betrachten das Dirichlet-Randwertproblem mit homogenen Randbedingungen:
- ,
Wir diskretisieren auf ein äquidistantes Punktegitter
mit konstanter Gittergröße .
Die Approximationen der Lösung in den Gitterpunkten werden mit bezeichnet.
Die zweiten Differenzenquotienten liefern die Approximation für nach dem Stern-Schema.
In den Randgitterpunkten werden die Werte von nach der homogenen Dirichlet-Randbedingung ersetzt:
,
.
Die unbekannten Approximationen der Lösung in den Gitterpunkten werden in einen langen Spaltenvektor eingeordnet, ebenso wie auch der Vektor der rechten Seite .
Insgesamt ergibt sich ein lineares Gleichungssystem
mit der blocktridiagonalen Matrix
mit Diagonalblock
und der Einheitsmatrix auf der Nebendiagonale.
Bei einem inhomogenen Randwertproblem sind die Randwerte der gesuchten Funktion durch Funktionen ungleich von Null folgendermaßen gegeben:
- linker Rand: ,
- rechter Rand:
- unterer Rand:
- oberer Rand: .
Dann tragen sie wie folgt zum Vektor der rechten Seite bei: .
Neumann-Randbedingungen
Ist der Fluss über die Ränder in Form der Richtungsableitungen der Funktion in der Richtung des äußeren Normalenvektoren gegeben, erfüllt die Neumann-Randbedingung:
Poisson-Gleichung mit Neumann-Randwertproblem
Die Approximationen für die inneren Gitterpunkte liefern die selben Stern-Approximationen wie die Dirichlet-Randbedingungen. Die Randbedingungen verändern lediglich einige Blöcke der tridiagonalen Matrix.
Seien die Ableitungen der unbekannten Funktion in Richtung der äußeren Normalenvektoren in den Randpunkten des rechteckigen Gebietes folgendermaßen gegeben:
- linker Rand:
- rechter Rand:
- unterer Rand:
- oberer Rand:
wobei
Speziell ist am Rand des Rechtecks wegen der äußeren Normalenvektoren oder .
Der Vektor der Unbekannten muss bei der Anwendung des einseitigen Differenzenquotieunten zur Berechnung der ersten Ableitungen am Rand um die Randwerte erweitert werden.
- linker Rand:
- rechter Rand:
- unterer Rand:
- oberer Rand:
Die Blöcke der blocktridiagonalen Systemmatrix : werden jeweils um die 0-te und (n+1)-te Zeile erweitert, die Blockzeilen von werden um die 0-te und (m+1)-te Blockzeile erweitert. Somit hat das lineare Gleichungssystem folgende Form:
mit Diagonalblock
und der Einheitsmatrix .
Dann Neumann-Randbedingungen tragen wie folgt zum Vektor der rechten Seite bei: .
Dirichlet
Implemetierung von inhomogenen Dirichlet-Randbedingungen in die inhomogene, stationäre Poissongleichung.
Definiere Gebiet
# Endpunkte x_end = 10; y_end = 10; # Schrittweite n = 100; hx = x_end/(n+1); hy = hx; m = floor(y_end/hy)-1; # Gitter erstellen x = [hx:hx:x_end-hx]; y = [hy:hy:y_end-hy]; [X,Y] = meshgrid(x,y);
Dirichlet RB & Diffusionskoeffizient
rand_links = -0.01; rand_rechts = 0.01; rand_oben = 0.01; rand_unten = -0.01; a = 1.0;
Systemmatrix erstellen
A = -4*eye(n*m,n*m)+diag(ones(n*m-1,1),1)+diag(ones(n*m-1,1),-1)+diag(ones(n*(m-1),1),n)+diag(ones(n*(m-1),1),-n); # -4 auf der Hauptdiagnonalen # 1 auf der 1., -1., n-ter und -n-ter Nebendiagonale # Korrektur von A for i=1:(m-1) A(i*n+1,i*n) = 0; A(i*n,i*n+1) = 0; endfor; # lösche jeden (i*n)-ten Eintag aus 1. und -1. Nebendiagonale A = (a/hx^2)*A;
Dirichlet RB in Funktion der rechten Seite einbetten
for i=1:n
for j=1:m
f(j,i)=1*Quellfunktion(X(j,i),Y(j,i));
# Dirichlet Randbedingungen
if i==1 f(j,i) = Quellfunktion(X(j,i),Y(j,i))+rand_oben*a/hx^2; endif
if i==n f(j,i) = Quellfunktion(X(j,i),Y(j,i))+rand_unten*a/hx^2; endif
if j==1 f(j,i) = Quellfunktion(X(j,i),Y(j,i))+rand_links*a/hx^2; endif
if j==m f(j,i) = Quellfunktion(X(j,i),Y(j,i))+rand_rechts*a/hx^2; endif
endfor
endfor
# Ecken haben Mittelwert aus anliegenden Rändern
#links-oben
f(1,1) = Quellfunktion(X(1,1),Y(1,1))+rand_links*a/hx^2+rand_oben*a/hx^2;
#links-unten
f(1,n) = Quellfunktion(X(1,n),Y(1,n))+rand_links*a/hx^2+rand_unten*a/hx^2;
#rechts-oben
f(m,1) = Quellfunktion(X(m,1),Y(m,1))+rand_rechts*a/hx^2+rand_oben*a/hx^2;
#rechts unten
f(m,n) = Quellfunktion(X(m,n),Y(m,n))+rand_rechts*a/hx^2+rand_unten*a/hx^2;
Lösung der stationären DGL
# f transponieren und als Vektor schreiben
f2 = -1*reshape(f',n*m,1);
# Lösung u bestimmen
u = (A\f2);
# u wieder als Matrix schreiben und wieder transponieren
u_matrix = reshape(u,n,m)';
==== Grafische Ausgabe ====
# Quellfunktion plotten
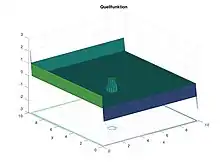
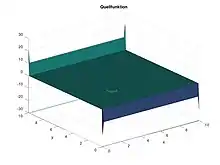
figure(1)
surfc(X,Y,f)
title("Quellfunktion")
xlabel("x")
ylabel("y")
test=["Fig_", num2str(1), ".jpg"]
saveas(1, test)
# Lösung plotten
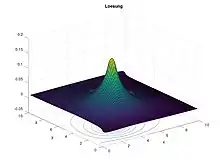
figure(2)
surfc(X,Y,u_matrix)
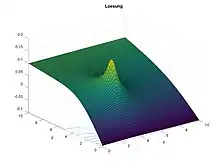
title("Loesung")
xlabel("x")
ylabel("y")
test=["Fig_", num2str(2), ".jpg"]
saveas(2, test)
Neumann mit Dirichlet RB
Definiere Gebiet
# Endpunkte x_end = 10; y_end = 10; # Schrittweite n = 100; hx = x_end/(n+1); hy = hx; m = floor(y_end/hy)-1; # Gitter erstellen x = [hx:hx:x_end-hx]; y = [hy:hy:y_end-hy]; [X,Y] = meshgrid(x,y);
Dirichlet RB & Diffusionskoeffizient
rand_oben = 0.01; rand_unten = -0.01; a = 1.0;
Systemmatrix erstellen
A = -4*eye(n*m,n*m)+diag(ones(n*m-1,1),1)+diag(ones(n*m-1,1),-1)+diag(ones(n*(m-1),1),n)+diag(ones(n*(m-1),1),-n); # -4 auf der Hauptdiagnonalen # 1 auf der 1., -1., n-ter und -n-ter Nebendiagonale # Korrektur von A for i=1:(m-1) A(i*n+1,i*n) = 0; A(i*n,i*n+1) = 0; endfor; # lösche jeden n-ten Eintag aus 1. und -1. Nebendiagonale A = (a/hx^2)*A; # Integriere Neumann Randbedingungen in A linke und rechte Seite # Normalenableitung bzw. Gradient*Normalenvektor am Rand ist mit 0 vorgegeben # In x-Richtung gilt: (du/dn_vec) = 0 --> (du/dx) = 0 # Das wird in der Physik häufig so gewählt for i=1:(m-2) vector_up = zeros(1,n*m); vector_up(n*i+1) = a/hx; vector_up(n*i+2) = -a/hx; vector_down = zeros(1,n*m); vector_down(n*i+n-1) = -a/hx; vector_down(n*i+n) = a/hx; A(i*n+1,:) = vector_up ; A(i*n+n,:) = vector_down ; endfor
Dirichlet RB in Funktion der rechten Seite einbetten
for i=1:n
for j=1:m
f(j,i)=1*Quellfunktion(X(j,i),Y(j,i));
#Dirichlet Randbedingung oben und unten implementieren:
if j==1
f(j,i)=1*Quellfunktion(X(j,i),Y(j,i))+rand_oben*a/hx^2;
elseif j==m
f(j,i)=1*Quellfunktion(X(j,i),Y(j,i))+rand_unten*a/hx^2;
endif
endfor
endfor
# Ecken haben Mittelwert aus anliegenden Rändern
f(1,1) = Quellfunktion(X(j,i),Y(j,i))+2*rand_oben*a/hx^2;
f(1,n) = Quellfunktion(X(j,i),Y(j,i))+2*rand_oben*a/hx^2;
f(m,1) = Quellfunktion(X(j,i),Y(j,i))+2*rand_unten*a/hx^2;
f(m,n) = Quellfunktion(X(j,i),Y(j,i))+2*rand_unten*a/hx^2;
Lösung der stationären DGL
# f transponieren und als Vektor schreiben
f2 = -1*reshape(f',n*m,1);
# Lösung u bestimmen
u = (A\f2);
# u wieder als Matrix schreiben und wieder transponieren
u_matrix = reshape(u,n,m)';
==== Grafische Ausgabe ====
# Quellfunktion plotten
figure(1)
surfc(X,Y,f)
title("Quellfunktion")
xlabel("x")
ylabel("y")
test=["Fig_", num2str(1), ".jpg"]
saveas(1, test)
# Lösung plotten
figure(2)
surfc(X,Y,u_matrix)
title("Loesung")
xlabel("x")
ylabel("y")
test=["Fig_", num2str(2), ".jpg"]
saveas(2, test)
A5 Instationäre Diffusion mit Reaktionsterm
Ziel ist die zeitlich-räumliche Modellierung der Epidemie auf einem Rechteckgebiet mit homogenen Neumann-Randbedingungen , nicht-konstantem Diffusionskoeffizient , geographisch differenzierter Bevölkerungsdichtefunktion , der anhand der RKI Daten festgestellten zeitabhängigen Infektionsraten und einer Anfangslösung.
zeitdiskretisierte Lösung mit explizitem Euler-Verfahren
Die Lösung lässt sich mithilfe des expliziten Euler-Verfahrens mit der Konvergenzordnung 1 durch das Ersetzten von durch den
Vorwärtsdifferenzenquotienten approximieren.
Mit erhält man die Berechnungsformel
mit dem Startvektor
Die Lösung berechnet sich iterativ. Wegen der Stabilität muss die Schrittweite hinreichend klein gewählt werden.
raumabhängiger Diffusionskoeffizient
Um die räumliche Epidemieausbreitung regional zu differenzieren, wird der nicht-konstante Diffusionskoeffizient in Abhängigkeit von der Populationsdichte gestellt:.
Die homogene Neumann Randbedingung in diesem Fall lautet:
- .
Mit Anwendung der einseitigen Differenzenquotienten und der Bezeichnung erhält man
- am linken Rand des Rechtecks:
- ,
- am rechten Rand des Rechtecks:
- ,
- am unteren Rand des Rechtecks:
- ,
- am oberen Rand des Rechtecks:
- .
Das Approximationsschema für den Diffusionsterm
wird mit zweifacher Anwendung des zentralen Differenzenquotienten mit halber Schrittweite in den inneren Gitterpunkten hergeleitet:
- .
- .
Herleitung des zweiten Differenzenqutiont bez. x (analog bez. y):
- ,
- ,
Approximationsschema
Unter der Anwendung der Approximation der Randbedingungen und des Diffusionsterms erhalten wir folgende Systemmatrix
wobei
und .
In den obigen Matrizen kann man die Zwischenwerte der Funktion c durch die Mittelwerte ersetzen:
Die Funktion der rechten Seite berechnet sich folgendermaßen:
- für die Berechnung der Dichte der kumulierten Infizierten, oder
- für die Berechnung der Dichte der aktuellen Infizierten nach dem Kompartiment-Prinzip
Es ergibt sich folgendes System von gewöhnlichen Differentialgleichungen für die Reaktionsdiffusionsgleichung:
Gemeinsam mit der Anfangsbedingung
definiert sich das Anfangswertproblem für Reaktionsdiffusionsgleichung.
a) FDM-Verfahren implementieren zur Lösung der Reaktionsdiffusionsgleichung für die zeitlich-räumliche Modellierung einer Epidemie auf einem rechteckigen Gebiet: kummulierte Infizierte
Voraussetzungen:
- homogene Neumann-Randbedingungen
- konstanter Diffusionskoeffizienten c(x) = c (in Teilaufgabe a) und b), in c) abhängig von Bevölkerungsdichte)
- konstante, aber realistische Bevölkerungsdichtefunktion B(x)
- Anfangslösung als Startvektor definieren --> weitere Zeitschritte werden daraus berechnet
- Funktion des Reaktionsterms: Bevölkerungsdichte geographisch differenzieren als Funktion der Raumvariablen:
Berechnung der Dichte der kummuliert Infizierten:
:
Implementierung:
%Funktionsparameter: N-Anzahl von Gitterpunkten in x Richtung (0-te und N-te Index entsprechen den Rändern) %definiere Gebiet
Startparameter der Zeit und des Gebiets festlegen:
xend = 14; #in 22,5km yend = 15; #in 22,5km N = 100; T = 130; #Zeitintervall in Tagen delta_t = 0.03; #Zeitschritt b_mittel = 0.237; #mittlere Bevölkerungsdichte Deutschland hx = xend/(N+1); hy = hx; h = hx; M = floor((yend/hy))-1; Nr_timesteps = floor (T/delta_t); [x,y] = meshgrid(0:hx:xend,0:hy:yend); N = N+2; M = M+2;
Startparameter Reaktionsdiffusion festlegen
a = 0.1; # Diffusionskoeffizient w = 1./14; # Wechselrate
Bevölkerungsdichte: 1000 Menschen pro km^2
for i=1:N; for j=1:M; b(j,i) = Bd(1+floor(j/M*yend),1+floor(i/N*xend))*22.5^2; endfor; endfor; b = flipud(b); B=1*reshape(b',N*M,1); # als Vektor darstellen
Systemmatrix erstellen:
Vec1=ones(N*M,1); % erzeugt vektor der Laenge N*M
BB=diag(-4.*Vec1,0)+diag(ones(N*M-1,1),1)+ diag(ones(N*M-1,1),-1)+diag(ones(N*(M-1),1),N)+diag(ones(N*(M-1),1),-N);
# -4 auf der Hauptdiagnonalen
# 1 auf der 1., -1., nter und -nter Nebendiagonale
# Korrektur der Matrix (Blockdiagonalität) for i=1:(M-1) BB(i*N+1,i*N)=0;BB(i*N,i*N+1)=0; endfor # Matrix mit Diffkoeff/h^2 multiplizieren BB=BB*a/hx^2;
Neumann-Randbedingungen:
# Neumann RB für y: a* partial_y u=0 - einzelne Blöcke der Matrix ersetzen block=diag((a/hx)*ones(N,1)); %unten BB(1:N,1:N)=-block; BB(1:N,N+1:2*N)=block; %oben BB(N*(M-1)+1:N*M,N*(M-1)+1:N*M)=-block; BB(N*(M-1)+1:N*M,N*(M-2)+1:N*(M-1))=block;
# Neumann RB für x: a* partial_x u=0 - einzelne Zeilen der Matrix ersetzen for i=1:(M-2)% bei full neumann 0:(M-1) vector_up=zeros(1,N*M); vector_up(N*i+1)=a/hx; vector_up(N*i+2)=-a/hx; vector_down=zeros(1,N*M); vector_down(N*i+N)=a/hx; vector_down(N*i+N-1)=-a/hx; BB(i*N+1,:) =-vector_up ; BB(i*N+N,:) =-vector_down ; endfor
Tridiagonale Matrix plotten:
# plot Blocktridiagonale Matrix
figure (11);
spy (BB);
xlabel("Spalten");
ylabel("Zeilen");
zlabel("Matrix B");
title ("Systematrix B");
Startkonzentration der kummulierten Infizierten:
# 25.4 Infizierte *10 for i=1:N; for j=1:M; initial(j,i)=initialfunction(x(j,i),y(j,i))/1000 *10; endfor; endfor;
Reaktionsterm für die kummulierten Infizierten:
F=@(U_old,t) slowdown2(t)*U_old./B.*(B-U_old);
Lösungsschritt:
sol_old = 1*reshape(initial',N*M,1); # als Vektor schreiben
# Alle Zeitschritte lösen mit Reaktionsterm for i=1:Nr_timesteps i #zeitlich und räumliche diskretisierung #inhomogene, instationäre DDGL sol = sol_old + BB*sol_old*delta_t + F(sol_old,i*delta_t)*delta_t; sol_old=sol; matrix_solution(:,i)=sol; endfor
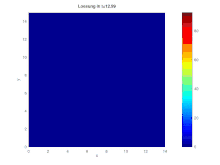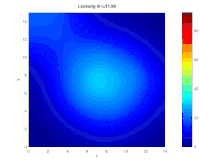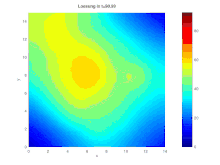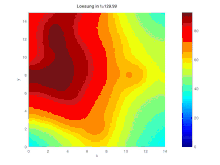
Plotten:
# jedes zehnte Bild plotten fig_index=floor([0.1:0.025:1]*Nr_timesteps); j=0;
for i=fig_index
sol_matrix=reshape(matrix_solution(:,i),N,M);% Matrix mit N(Zeilen)x M(Spalten)
sol_matrix=flipud(sol_matrix');
disp(['Figure ',num2str(i/fig_index(1))]);
j=j+1;
figure(j);
surfc(x,y,sol_matrix, "EdgeColor", "none")
colormap ("jet")
colorbar
axis([0 xend 0 yend -0.01 max(max(matrix_solution))])
caxis ([0 max(max(matrix_solution))])
#title (["Loesung in t=", num2str(delta_t*i), " I_{ges}=", num2str(sum(sum(sol_matrix)))]);
title (["Loesung in t=", num2str(delta_t*i)])
ylabel("y")
xlabel("x")
view(0,90)
%Optional: Speicherung der Bilder
test=["Fig_", num2str(j),".jpg"]
saveas(j, test)
endfor
#weiteres Bild mit Anfangsfunktion
figure(Nr_timesteps+1);
surfc(x,y,initial);
title ("Anfangslösung");
ylabel("y")
xlabel("x")
b) wie in a) nur für Dichte der aktuell Infizierten:
Implementierung: siehe oben bis Startkonzentration aktuell Infizierte:
# 3.24 Infizierte verteilt *1 # 32.4 Infizierte *10 for i=1:N; for j=1:M; initial(j,i)=initialfunction(x(j,i),y(j,i))/1000 *10; endfor; endfor;
Reaktiionsterm aktuell Infizierte:
FS=@(U_old_I, U_old_S,t) -slowdown2(t)*U_old_I./B.*U_old_S;
FI=@(U_old_I, U_old_S,t) slowdown2(t)*U_old_I./B.*U_old_S-w*U_old_I;
.gif)
Lösungsschritt:
sol_old_I = 1*reshape(initial',N*M,1); sol_old_S = B.-sol_old_I; # als Vektor schreiben
# Alle Zeitschritte lösen mit Reaktionsterm for i=1:Nr_timesteps i sol_I = sol_old_I + BB*sol_old_I*delta_t + FI(sol_old_I,sol_old_S,i*delta_t)*delta_t; sol_S = sol_old_S + BB*sol_old_S*delta_t + FS(sol_old_I,sol_old_S,i*delta_t)*delta_t; sol_old_I=sol_I; sol_old_S=sol_S; matrix_solution_I(:,i)=sol_I; matrix_solution_S(:,i)=sol_S; endfor
Plotten
c) Nicht-konstanter Diffusionskoeffizient
Ausbreitungsgeschwindigkeit abhängig von der Populationsdichte:
Entsprechende Diffusionsmatrix wird für die für die Diskretisierung von
angepasst.
Diffusionskoeffizient
Da wir beim Diffusionskoeffizienten auch halbe Schrittweiten berücksichtigen müssen, muss c auf 2N x 2M ausgedehnt werden. Die fehlenden Werte, werden durch die Mittelwerte der anliegenden Nachbarn approximiert.
Diffusionskoeffizient-Matrix bestimmen
# c=c(B): Diffusionskoeffizient abhängig von der Bevölkerungsdichte
function val = c_function(a,b,p,N,M)
c=zeros(2*M,2*N);
#b auf jeden zweiten Gitterpunkt von c verteilen
for i=1:N
for j=1:M
c(2*j,2*i)=a+p*b(j,i);
endfor
endfor
#Zwischenwerte bestimmen als Mitelwert der umliegenden
#Erste Zeile bestimmen
for i=1:N
c(1,2*i) = c(2,2*i);
endfor
for i=2:N
c(1,2*i-1) = 1/2*(c(1,2*i-2)+c(1,2*i));
endfor
#Erste Spalte bestimmen
for j=1:M
c(2*j,1) = c(2*j,2);
endfor
for j=2:M
c(2*j-1,1) = 1/2*(c(2*j-2,1)+c(2*j,1));
endfor
#rechte obere Ecke
c(1,1)=1/2*(c(1,2)+c(2,1));
#Zeilen auffüllen
for j=1:M
for i=1:N-1
c(2*j,2*i+1)=1/2*(c(2*j,2*i)+c(2*j,2*i+2));
endfor
endfor
for i=2:2*N
for j=1:M-1
c(2*j+1,i)=1/2*(c(2*j,i)+c(2*j+2,i));
endfor
endfor
val = c;
endfunction
Blöcke der Systemmatrix erstellen
Zunächst müssen die einzelnen Blöcke der Systemmatrix erstellt werden.
I_Eins Matrix
# I_0 Matrix erstellen
function y = I_Eins_Matrix(j,c,h,N,M)
I_Eins = zeros(N,N);
for i=1:N
I_Eins(i,i) = c(2*1,2*i);
endfor
y = I_Eins;
endfunction
I_M Matrix
# I_m+1 Matrix erstellen
function y = I_M_Matrix(j,c,h,N,M)
I_M = zeros(N,N);
for i=1:N
I_M(i,i) = c(2*M,2*i);
endfor
y = I_M;
endfunction
I_l_j Matrix
# Î_l_j Matrix erstellen
function y = I_l_j_Matrix(j,c,h,N,M)
I_l_j = zeros(N,N);
for i=2:N-1
I_l_j(i,i) = c(2*(j-1/2),2*i);
endfor
y = I_l_j;
endfunction
I_r_j Matrix
# Î_r,j Matrix erstellen
function y = I_r_j_Matrix(j,c,h,N,M)
I_r_j = zeros(N,N);
for i=2:N-1
I_r_j(i,i) = c(2*(j+1/2),2*i);
endfor
y = I_r_j;
endfunction
B_j Matrix
#Gibt uns die Matrix B_j aus
function y = B_j_Matrix(j,c,h,N,M)
# B_j Matrix erstellen
B_j = zeros(N,N);
#oberer linker Eintrag
B_j(1,1) = -h*c(2*j,2*1);
#unterer rechter Eintrag
B_j(N,N) = -h*c(j,N);
#untere Nebendiagonale
#oben rechts
B_j (N,N-1) = h*c(2*j,2*N);
for i=1:N-2
B_j(2+i-1,1+i-1) = c(2*j,2*(i+1/2));
endfor
#obere Nebendiagonale
#oben links
B_j(1,2) = h*c(2*j,2*1);
for i=2:N-1
B_j(1+i-1,2+i-1) = c(2*j,2*(i+1/2));
endfor
#Hauptdiagnonale
for i=2:N-1
B_j(i,i) = -(c(2*j,2*(i-1/2))+c(2*j,2*(i+1/2))+c(2*(j+1/2),2*i)+c(2*(j-1/2),2*i));
endfor
y = B_j;
endfunction
Systemmatrix aus Blöcken generieren
B_j Matrix
#Block-Tri-Diagonale Matrix
function val = BB_function(c,h,a,N,M)
BB=zeros(N*M,N*M);
#oben links
BB(1:N,1:N) = -h*I_Eins_Matrix(j,c,h,N,M);
#rechts daneben
BB(1:N,N+1:2*N) = h*I_Eins_Matrix(j,c,h,N,M);
#unten rechts
BB((M-1)*N+1:N*M,(M-1)*N+1:N*M) = -h*I_M_Matrix(j,c,h,N,M);
#links daneben
BB((M-1)*N+1:N*M,(M-2)*N+1:N*(M-1)) = h*I_M_Matrix(j,c,h,N,M);
#Hauptdiagonale
#laufvariable gilt für zeile und spalte, weil Hauptdiagonale
for j=2:M-1
BB((j-1)*N+1:j*N,(j-1)*N+1:j*N) = B_j_Matrix(j,c,h,N,M);
endfor
#linke Nebendiagonale
for j=1:M-2
BB(j*N+1:(j+1)*N, (j-1)*N+1:j*N) = I_l_j_Matrix(j,c,h,N,M);
endfor
#linke Nebendiagonale
for j=2:M-1
BB((j-1)*N+1:j*N,j*N+1:(j+1)*N) = I_r_j_Matrix(j,c,h,N,M);
endfor
# Matrix mit 1/h^2 multiplizieren
BB=BB/h^2;
val = BB;
endfunction
Durchführung
Startparameter festlegen
#---------------------------------------------------------------# #--STARTPARAMETER-ZEIT_UND_GEBIET---# xend = 14; %in 22,5km yend = 15; %in 22,5km N = 50 ; a = 0.01; %(konstanter Diffusionskoeffizient) T = 130 ; % Zeitintervall in Tagen) delta_t = 0.03; %(Zeitschritt) hx = xend/(N+1); hy = hx; h = hx; M = floor((yend/hy))-1; Nr_timesteps = floor (T/delta_t); [x,y] = meshgrid(0:hx:xend,0:hy:yend); N = N+2; M = M+2; #---------------------------------------------------------------# #--STARTPARAMETER-REAKTIONSDIFFUSION---# a = 0.007; # Diffusionskoeffizient w = 1./14; # Wechselrate c_basis = 0.3219; # Basisinfektionsrate b_mittel = 0.237; # 100 Menschen pro qkm p = 0.00001 ; # Proportionalitätsfaktor für Diffusionskoeffizient
Bevölkerungsdichte festlegen
# Bevölkerungsdichte (1000 Menschen pro h^2)
for i=1:N;
for j=1:M;
b(j,i) = Bd(1+floor(j/M*yend),1+floor(i/N*xend))*22.5^2;
endfor;
endfor;
b = flipud(b);
B=1*reshape(b',N*M,1);
# als Vektor darstellen
Anfangswert der Infizierten festlegen
#--STARTKONZENTRATION-INF--# # 28 Infizierte *10 for i=1:N; for j=1:M; initial(j,i)=initialfunction(x(j,i),y(j,i))/1000 *10; endfor; endfor;
Reaktionsterme
# kumulierte Infizierte #F=@(U_old,t) slowdown2(t)*U_old./B.*(B-U_old); # aktuell Infizierte FS=@(U_old_I, U_old_S,t) -slowdown2(t)*U_old_I./B.*U_old_S; FI=@(U_old_I, U_old_S,t) slowdown2(t)*U_old_I./B.*U_old_S-w*U_old_I;
Iterationen durchführen
# anfängliche Bevölkerungsdichte als Vektor schreiben sol_old_I = 1*reshape(initial',N*M,1); sol_old_S = B.-sol_old_I;
# anfängliche Basisinfektionsmatrix aufstellen c = c_function(a,b,p,N,M); # minimalen und maximalen Diffusionskoeffizienten ausgeben c_min = min(min(c)) c_max = max(max(c)) # Alle Zeitschritte lösen mit Reaktionsterm for i=1:Nr_timesteps i sol_I = sol_old_I + BB_function(c,h,a,N,M)*sol_old_I*delta_t + FI(sol_old_I,sol_old_S,i*delta_t)*delta_t; sol_S = sol_old_S + BB_function(c,h,a,N,M)*sol_old_S*delta_t + FS(sol_old_I,sol_old_S,i*delta_t)*delta_t; sol_old_I=sol_I; sol_old_S=sol_S; matrix_solution_I(:,i)=sol_I; matrix_solution_S(:,i)=sol_S; endfor
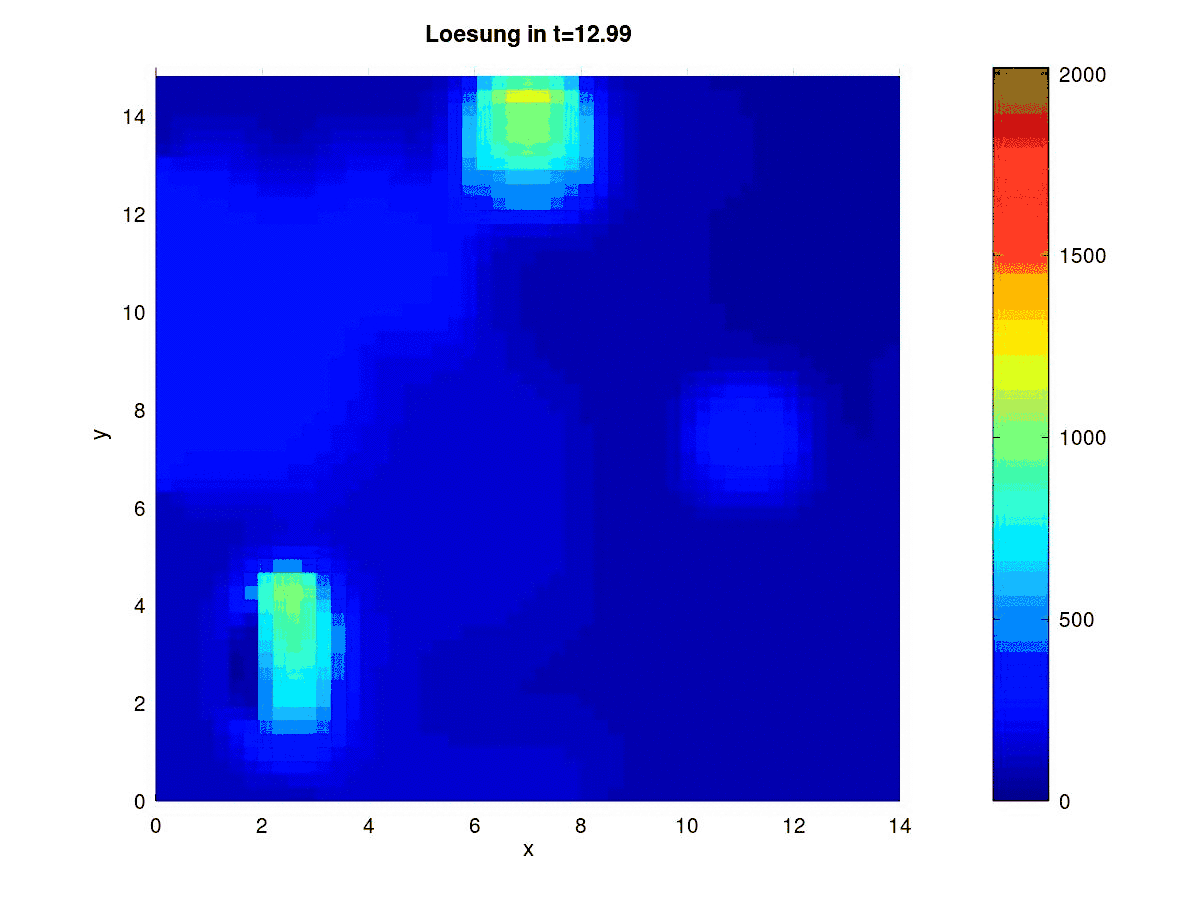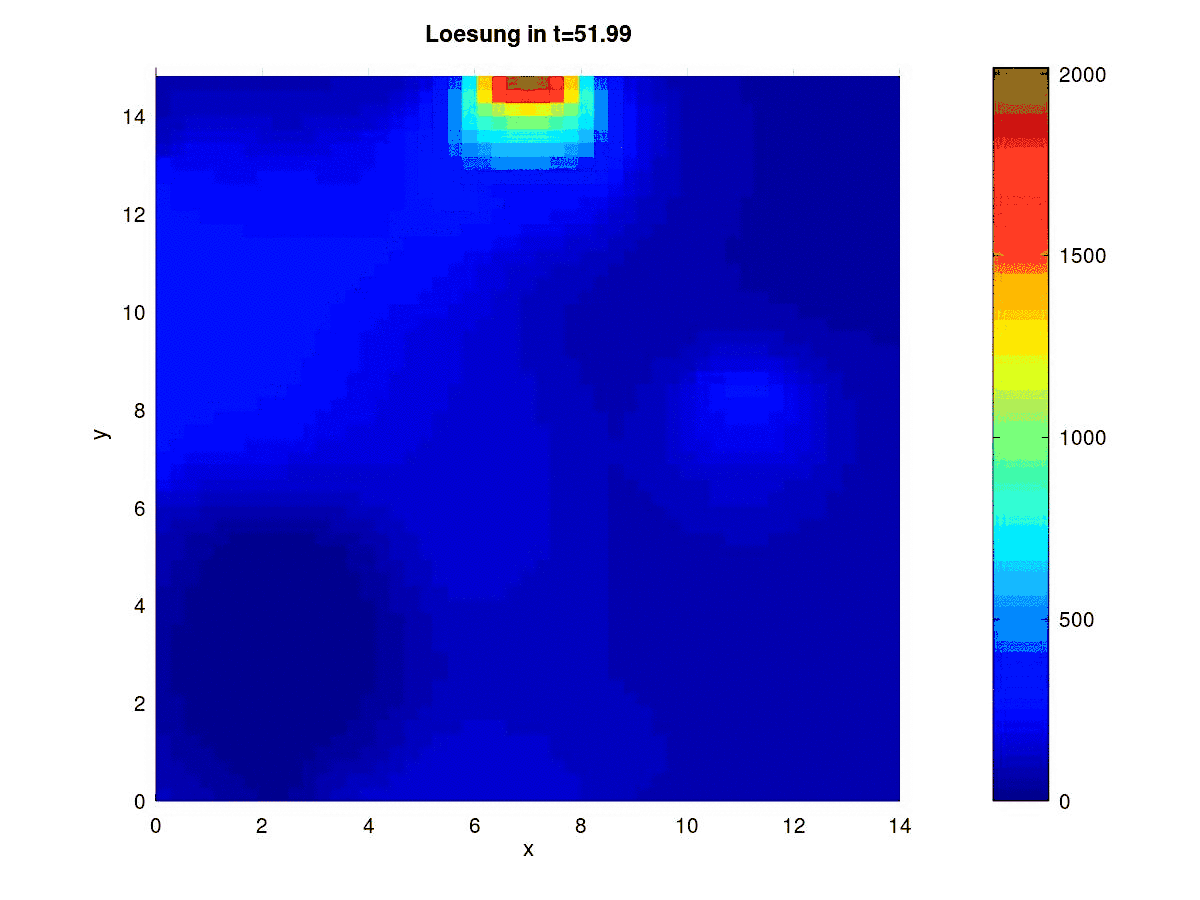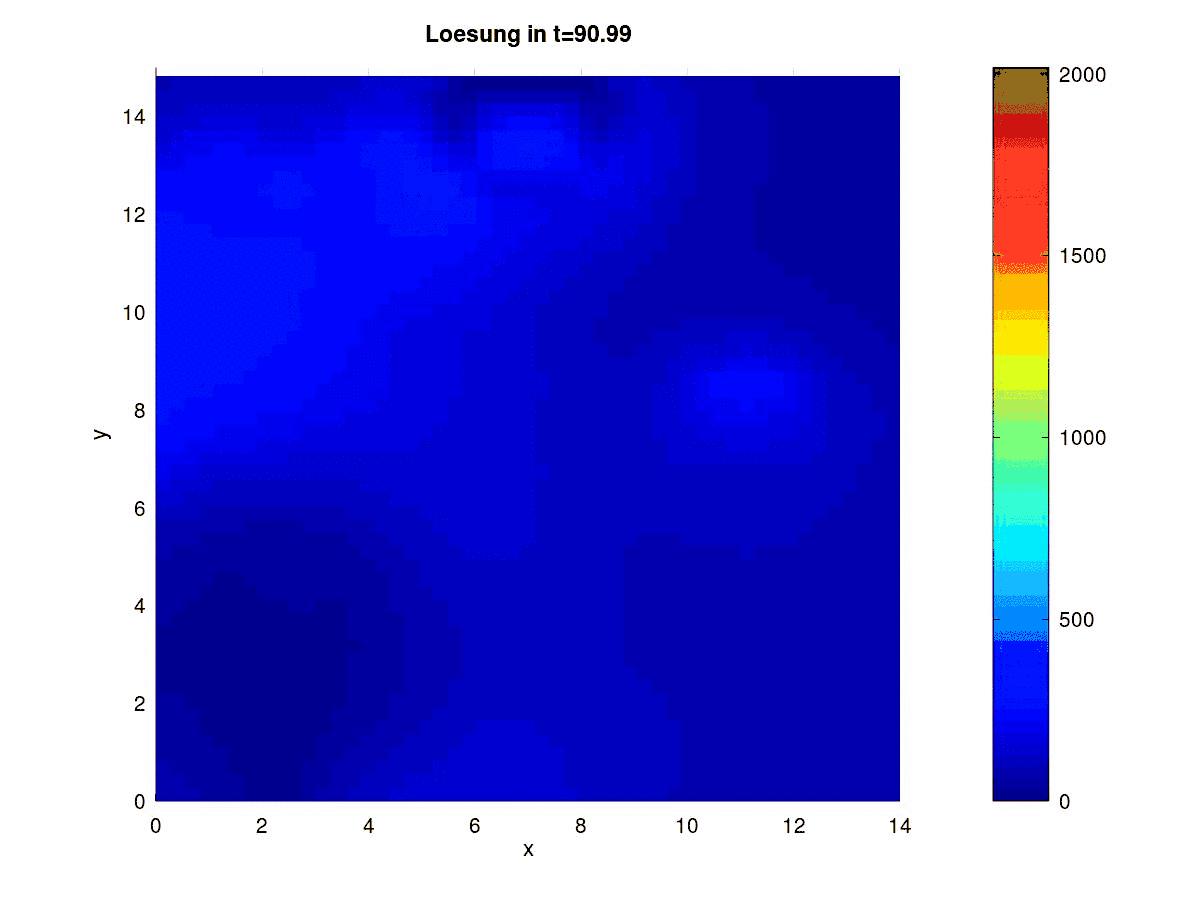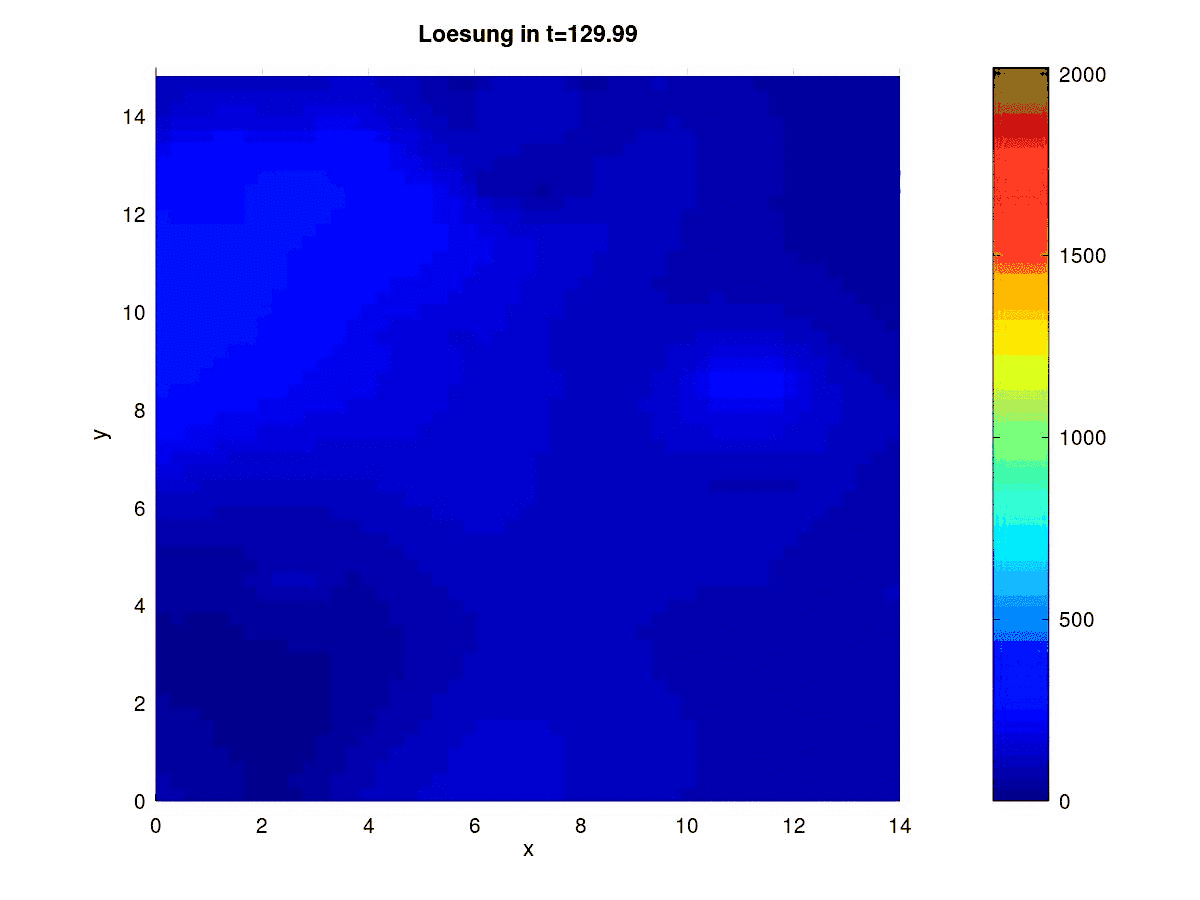
Graphische Ausgabe
fig_index=floor([0.1:0.025:1]*Nr_timesteps);
j=0;
for i=fig_index
sol_matrix=reshape(matrix_solution_I(:,i),N,M);% Matrix mit N(Zeilen)x M(Spalten)
sol_matrix=(sol_matrix');
disp(['Figure ',num2str(i/fig_index(1))]);
j=j+1;
figure(j);
surfc(x,y,sol_matrix, "EdgeColor", "none")
colormap ("jet")
colorbar
axis([0 xend 0 yend -0.1 max(max(matrix_solution_I))])
caxis ([0 max(max(matrix_solution_I))])
#title (["Loesung in t=", num2str(delta_t*i), " I_{ges}=", num2str(sum(sum(sol_matrix)))]);
title (["Loesung in t=", num2str(delta_t*i)])
ylabel("y")
xlabel("x")
view(0,90)
%Optional: Speicherung der Bilder
test=["Fig_", num2str(j),".jpg"]
saveas(j, test)
endfor
#weiteres Bild mit Anfangsfunktion
figure(Nr_timesteps+1)
surfc(x,y,initial)
title ("Anfangslösung")
ylabel("y")
xlabel("x")
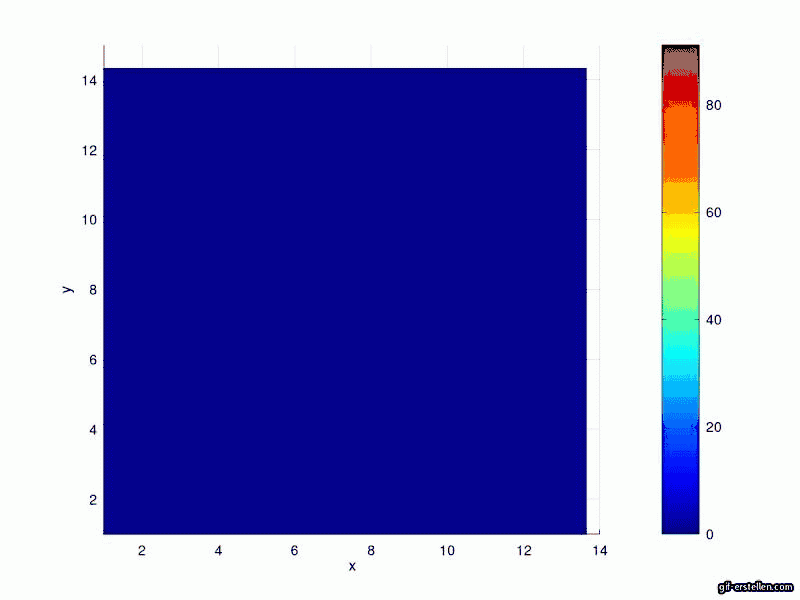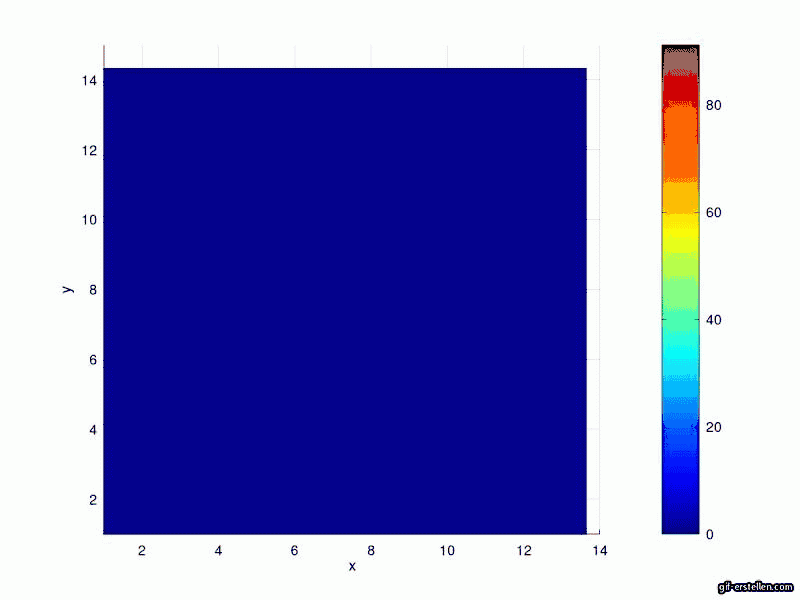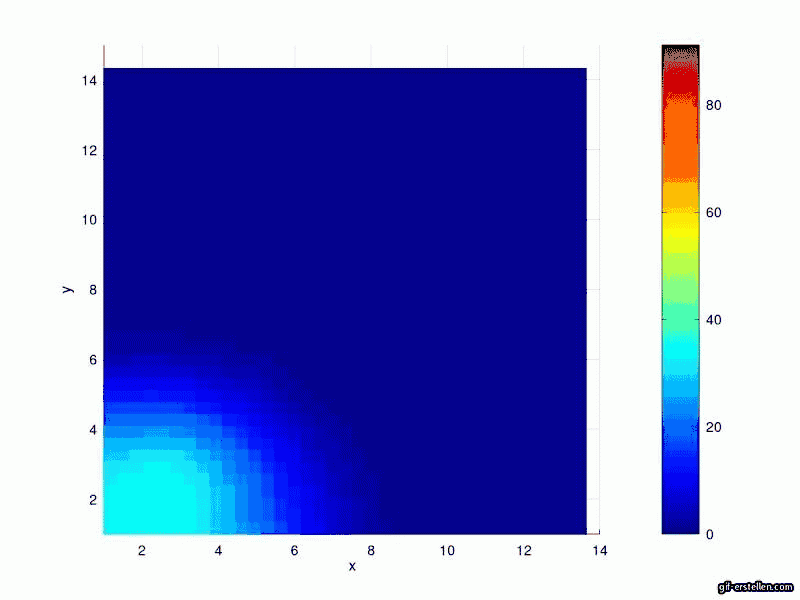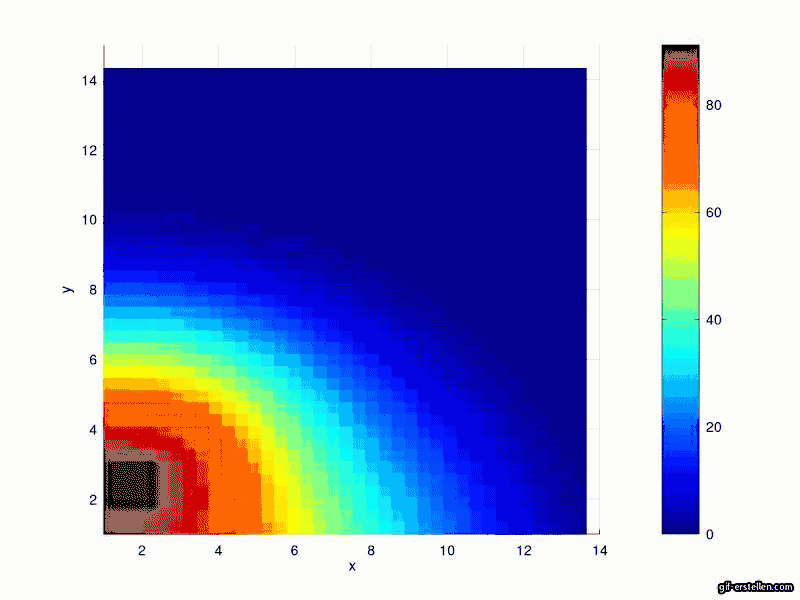
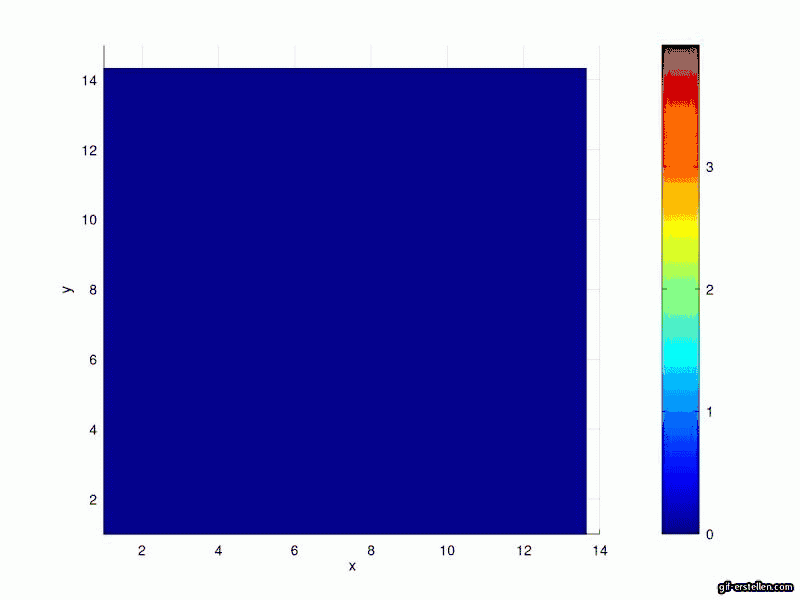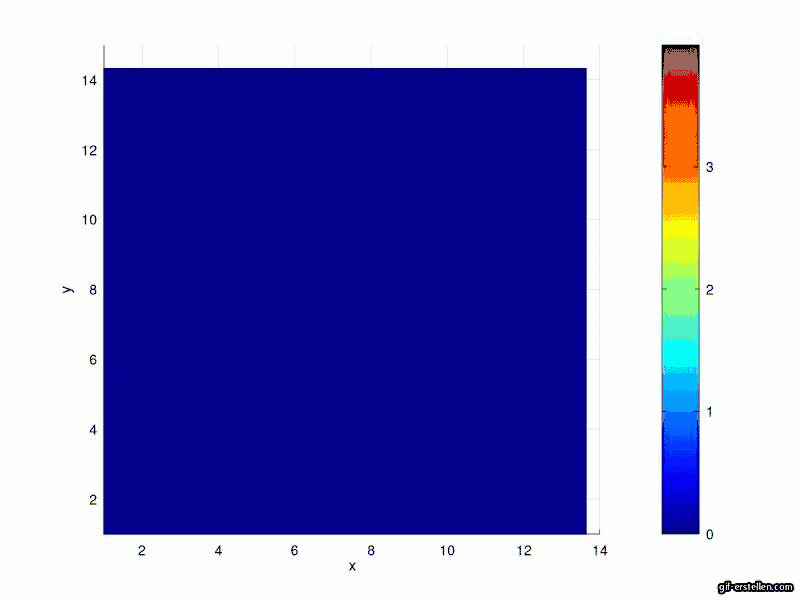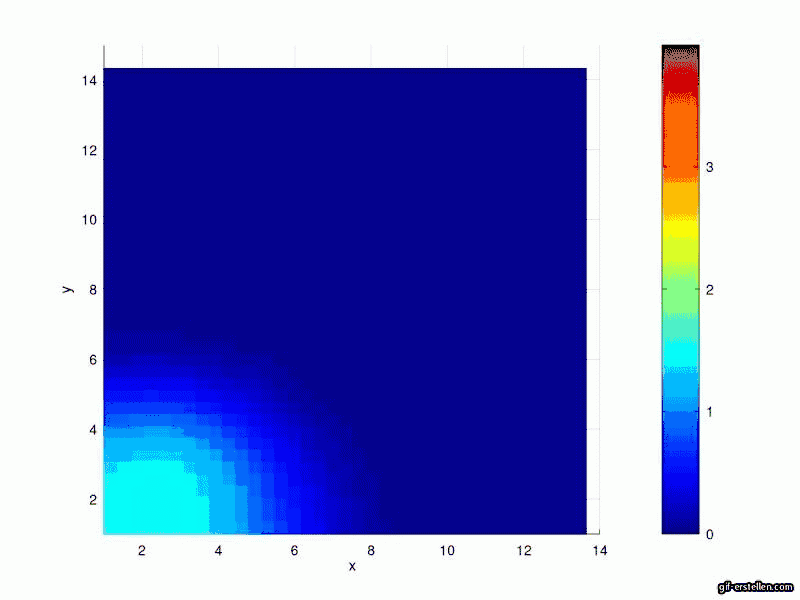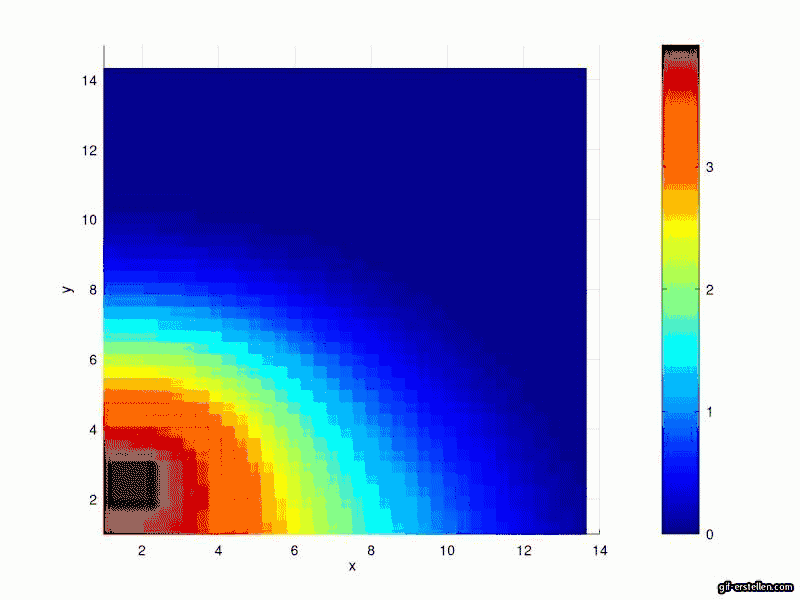
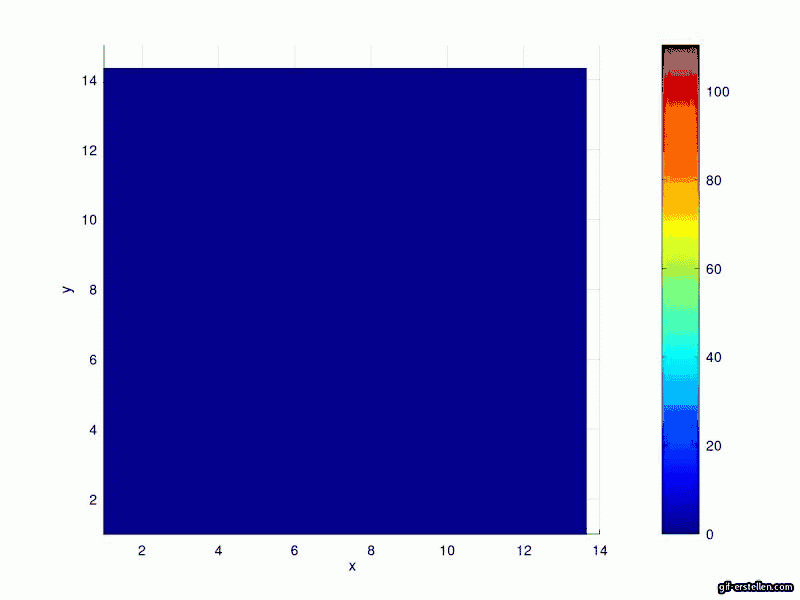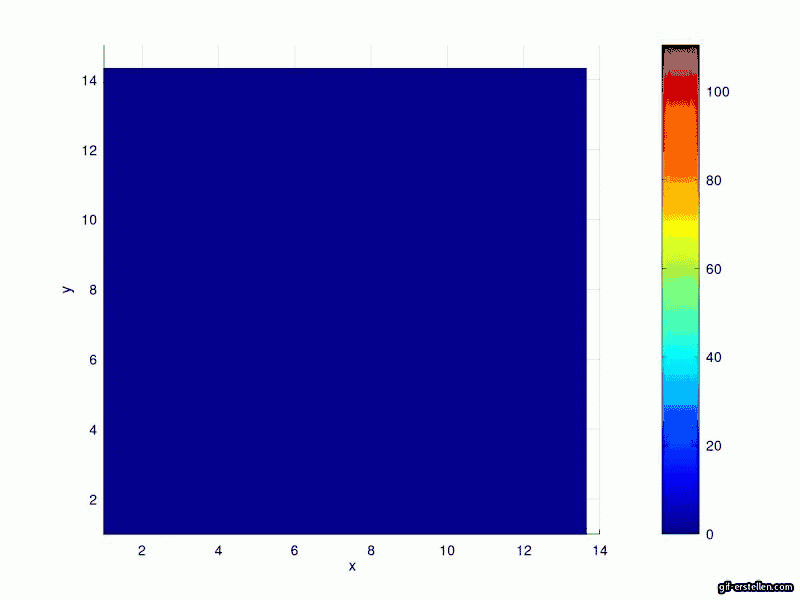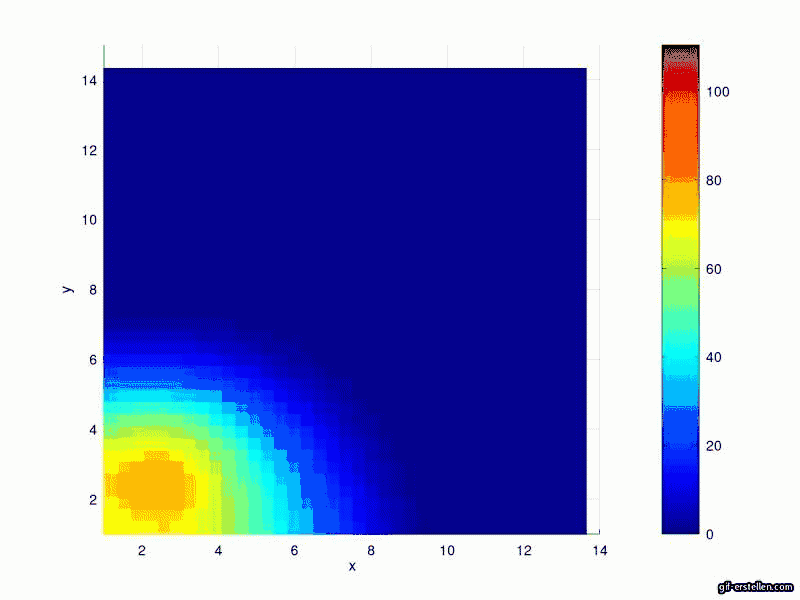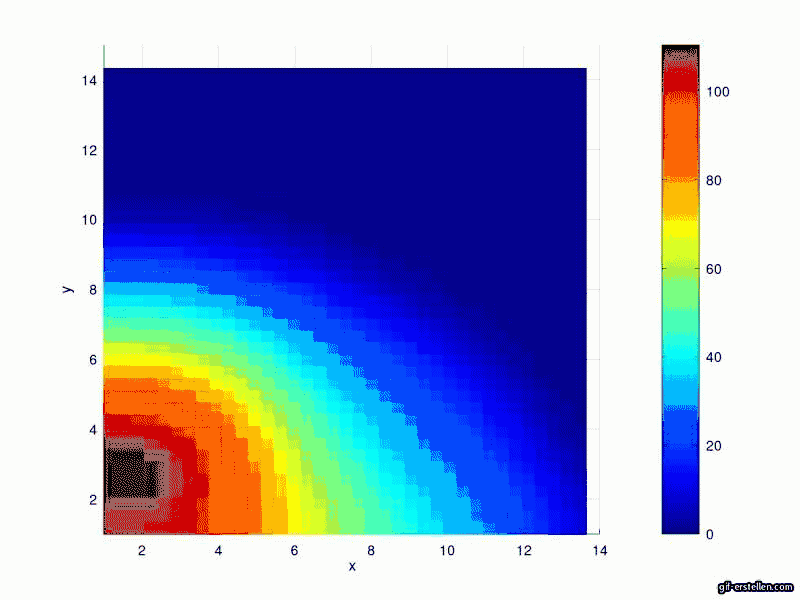
Ergebnisse
Aktuell InfizierteSusceptible
Skripte in Octave
Referenzieren Sie hier die von Ihnen verwendeten Skripte und Bibliotheken in Octave oder R/Studio
Literatur
Notieren Sie hier die von Ihnen verwendete Literatur
- Boto von Querenburg (2013) Mengentheoretische Topologie - Springer-Lehrbuch
.gif)
.gif)
.gif)